TGF-β/SMAD 信号通路在生物系统中对细胞生长、增殖、分化和迁移的调节起着至关重要的作用。同时,TGF-β/SMAD 信号通路的空间、时间和动态变化能直接调节各种细胞功能,从而影响动物发育和疾病。该通路的信号转导起始于配体TGF-β,通过激活下游转录因子SMAD2 或者 SMAD3来调节各种不同细胞状态特异性基因转录。目前已知TGF-β 家族受体和SMADs的蛋白稳定性由 Smurf E3 泛素连接酶和 USP4/11/15 去泛素化酶共同调节。由于TGF-β/SMAD信号通路复杂的空间、时间动态变化和调控不同生物学效应的复杂性,目前对其转录因子SMAD翻译后修饰仍未完全了解。因此深入研究SMAD翻译后的修饰对了解TGF-β信号通路在人类疾病中的作用机制,并为其临床靶向治疗提供有效的分子理论依据意义重大。
2022年1月3日,湖南大学生命医学交叉研究院周俊课题组与德国法兰克福大学承辛来课题组联合德国癌症研究中心和海德堡大学的研究人员在Journal of Cell Biology杂志上发表了题为 pVHL-mediated SMAD3 degradation suppresses TGFß signalling的研究论文。该研究发现了 Von-Hippel-Lindau-肿瘤抑制因子 (pVHL)作为E3泛素连接酶能调控 SMAD3泛素化和降解。这种在人体细胞和果蝇组织中对SMAD3 的负调控作用表明 pVHL在调节 TGF-β/SMAD信号通路中的保守作用,同时在维持组织稳态和动物发育中发挥着重要的作用。

TGF-β/SMAD 信号通路主要调控细胞增殖、迁移和组织发育。 该信号通路的失调与发育障碍和许多疾病息息相关。因此,有需要更深入地了解 TGF-β/SMAD 信号通路的动态翻译后修饰的分子机制。作者先前的工作已表明的靛玉红衍生物抑制剂 (E738) 会影响 SMAD3蛋白的稳定性【1】。在本研究中,他们结合蛋白质组学和 siRNA 筛选的方法并确定了E3 泛素连接酶 pVHL 与 SMAD3 泛素化和降解有关。为了研究 pVHL 如何与 SMAD3 相互作用并对其进行降解,他们进行了一系列生化分子实验分析。他们的实验证明了保守的 Mad 同源结构域 (MH2) 中的 LxLxxP 序列对于 pVHL 介导的 SMAD3 降解是不可或缺的。利用人类细胞模型,作者们的结果表明 pVHL 对 TGF-β/SMAD3 信号通路诱导的细胞迁移作用的影响呈负相关性。此外,在发育过程中的果蝇翅膀组织中,pVHL 反向调节 SMAD3 介导的细胞增殖、组织生长和发育模式形成。 目前已知TGF-β/SMAD 信号通路与多种人类疾病有关。如果在人类疾病中也能发现 pVHL 对 TGF-β/SMAD 信号通路的负调控作用,那将可以利用pVHL 作为治疗 TGF-β/SMAD 信号通路相关疾病(如自身免疫性疾病,纤维化和癌症)的药物靶点。
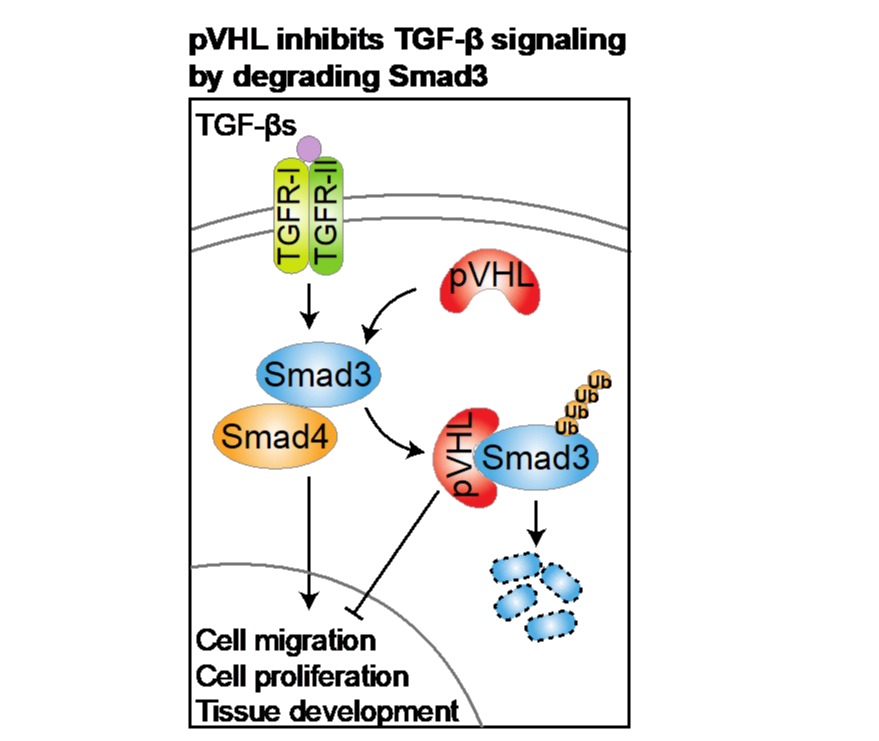
pVHL通过降解SMAD3来调节TGF-β信号通路的活性
湖南大学周俊教授与德国海德堡大学Yasamin Dabiri博士为本文共同第一作者,法兰克福大学的承辛来教授为本文的通讯作者。
湖南大学生命医学交叉研究院周俊课题组主要致力于利用动物模型研究信号通路与发育、菌群与宿主互作、干细胞与组织代谢、衰老等重要的热点领域。其相关成果主要发表在Journal of Cell Biology (2022),Developmental Cell(2021),Cell Chemical Biology(2021),PNAS (2020),Nature Communications (2017)等杂志上。现由于课题需要招聘助理教授1名和博士后1-2名,实验室常年招收硕士和博士研究生,请有意者将简历发送至 junzhou82@yahoo.com。 欢迎访问实验室PI网页了解详细信息 (http://smyjy.hnu.edu.cn/info/1135/1365.htm)。
原文链接:https://doi.org/10.1083/jcb.202012097
1. Cheng, X., H. Alborzinia, K.-H. Merz, H. Steinbeisser, R. Mrowka, C. Scholl, I. Kitanovic, G. Eisenbrand, and S. Wölfl. 2012. Indirubin derivatives modulate TGFβ/BMP signaling at different levels and trigger ubiquitin-mediated depletion of nonactivated R-Smads. Chem. Biol. 19:1423–1436.