RNA甲基化修饰在真核生物的基因表达调控中发挥着重要作用,其中,N6-甲基腺苷(m6A)是常见于信使RNA中的一种修饰,已被证实广泛参与基因的复制、转录、翻译,能够影响细胞的各种生理活动和功能 [1]。此外,m6A的调节异常与各种疾病的发生发展有着十分紧密的关系(如癌症、心血管疾病和神经系统疾病等)。
METTL3-METTL14甲基转移酶复合物是负责催化m6A的关键元件,包括催化亚基METTL3和辅助亚基METTL14,以及多个支架蛋白WTAP、ZC3H13和RBM15等 [2]。其中,METTL3起主要催化功能,METTL14维持结构完整与RNA结合。研究表明, METTL3在多种癌症中异常表达,如:在急性白血病、肝癌、胃癌、肺癌,结直肠癌等。并在多种癌症发挥致癌因子的作用,是表观遗传最热门的靶点之一 [3]。2021年,英国STORM Therapeutic公司开发了首个METTL3小分子抑制剂STM2457用于白血病的治疗,但其在实体瘤中的效果尚不可知 [4]。因此急需开发新型高效的抑制剂。多肽药物具有高度特异性和良好的安全性,特别是能够针对传统认为不可成药的蛋白蛋白互作区域进行药物开发,因此开发METTL3多肽抑制剂就具有极大的研究价值。
近日,湖南大学生命交叉研究院史俊峰和张定校课题组在Angewandte Chemie International Edition发表了一篇题为A Stapled Peptide Inhibitor Targeting the Binding Interface of N6-Adenosine-Methyltransferase Subunits METTL3 and METTL14 for Cancer Therapy的研究论文。该研究基于METTL3-METTL14蛋白结构,设计了靶向METTL3的多肽抑制剂RM3。进一步优化获得更高活性的订书肽抑制剂RSM3,该多肽具有广谱的抑癌特性,能够杀伤多种METTL3高表达的肿瘤细胞。同时在多种肿瘤模型中,RSM3显示出优异的抗肿瘤效果。

该论文基于METTL3-METTL14 (PDB: 5IL1[5])的晶体结构,在蛋白之间的两个相互作用界面(interface loop,residues 462-479), helix a2,residues 435-447)和一个底物催化中心(residues 534-541)中鉴定和设计了一系列多肽序列,并结合穿膜肽R9用于细胞内递送。通过体外酶活力测定、细胞存活、亲和力测定以及多肽pull down等实验筛选获得一段有生物活性的多肽RM3(源于residues 435-447),具有靶向METTL3、抑制METTL3酶催化活性和抑制癌细胞生长迁移等功能。同时,为了保持多肽的a二级结构和提高稳定性,进一步构建了订书肽化的多肽抑制剂RSM3(A)。
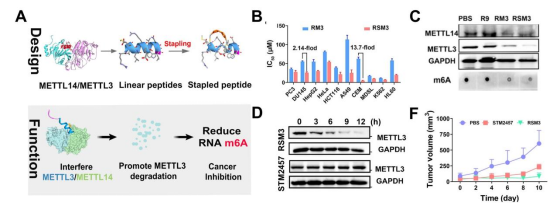
进一步细胞实验发现,该多肽可有效的降低细胞的mRNA甲基化水平,还能抑制多种癌细胞的生长和引起细胞凋亡,显示出较强的广谱抑癌性(B)。机制研究发现多肽抑制剂处理的细胞中METTL3表达量显著下降(C),推测其可引起METTL3的降解。进一步研究发现,该多肽主要通过蛋白酶体途径引发METTL3降解,而STM2457并未引起METTL3的蛋白含量下降(D),说明多肽抑制剂RSM3具有不同的作用机制。最终,在小鼠前列腺癌和胰腺癌等肿瘤模型中,RMS3等多肽抑制剂展现出较好的抑瘤能力,并具有较好的安全性(F)。此外,在活体水平RSM3的效果也略优于STM2457。以上结果说明该多肽具有较好的应用潜力。
湖南大学生命医学交叉研究院博士生李增辉和冯宇晴为本论文的共同第一作者,史俊峰和张定校教授为共同通讯作者。据悉,该抑制剂与PD-1联用还被2024年美国临床肿瘤学会年会ASCO(全球规模最大的、学术水平最高、最具权威的临床肿瘤学会议)收录,这也是湖大首次有工作入选ASCO。
原文链接:https://doi.org/10.1002/anie.202402611
参考文献
[1] Y. Fu, D. Dominissini, G. Rechavi, et al., Nat. Rev. Genet., 15 (2014) 293-306.
[2] J. Liu, Y. Yue, D. Han, et al., Nat. Chem. Biol., 10 (2013) 93-95.
[3] C. Zeng, W. Huang, Y. Li, et al., J. Hematol. Oncol., 13 (2020) 117.
[4] E. Yankova, W. Blackaby, M. Albertella, et al., Nature, 593 (2021) 597-+.
[5] X. Wang, J. Feng, Y. Xue, et al., Nature, 534 (2016) 575-578.